就像北极熊有厚厚的皮毛能扛住刺骨寒冷,蝙蝠用回声定位在漆黑中自由翱翔一样,每种细菌也都有自己的“绝活”,帮助它们在独特的环境中生存。比如,有些细菌能耐辐射,有些能制造冰核来“欺负”植物,还有一种生活在海洋藻类里的细菌,特别擅长“混搭”自己的DNA,频率高到让其他细菌望尘莫及。这种“混搭”能产生化学多样性,为宿主藻类提供防御武器。但要找出这些“绝活”背后的关键基因,就像是大海捞针——细菌基因组庞大,变异频繁,怎么才能精准锁定那些“特殊玩家”呢?以往方法多关注“孤儿基因”(那些独一无二的基因),却忽略了很多“变身”后的常见基因,它们才是真正赋予细菌超能力的功臣。
2025年10月29日,北京大学李志远和杨晓静团队在mBio杂志上发表了研究论文“A phylogeny-based method in detecting the species-specialized genes in microbes and its application to a marine symbiont bacterium”。研究团队开发了一种全新的计算机方法,能无偏倚地从细菌基因组中挖出那些“偏离常规”的基因变体。他们以一种海洋共生细菌Ca. Endoriftia kahalalidifaciens(简称cEK)为例,这种细菌像个寄居蟹一样住在海洋藻类Bryopsis sp.里面,帮助藻类制造防御化合物,并以比其它细菌高出数量级的重组能力产生防御化合物的多样性。团队发现,cEK的“混搭高手”能力来自几个关键基因(如ruvA/B/C、recG和ligA)的特殊变体,并在实验室细菌E. coli中验证,这些变体能大幅提升DNA“混搭”效率。
研究对象:一个疯狂重组的共生菌
研究的主角是一种生活在羽藻体内的共生细菌cEK,它是羽藻的“化学护卫队”,能制造复杂多样的防御化合物。之前我们发现,它的DNA重组速度高得离谱,把自己的防御体系像搭积木一样快速拼装重组,迅速创造出新的化合物组合。这种能力的分子基础尚未被发掘。
新计算方法:从“系统发育树”里找出异类
演化主导着基因的变迁,不同物种的基因会随着演化的系统发育树而产生规律性的变化,如果某个基因在某个物种中“偏离常规”,那它很可能是赋予该物种独特功能的关键。
研究团队收集了197种相关细菌的完整基因组数据,发明了“系统发育偏差分数”(PD分数):如果某个基因的序列在cEK中“独树一帜”,偏离了预期模式,就很可能是个“特殊基因”。他们计算了cEK的619个基因组,选出高分基因进行分析,结果显示这些基因多集中在DNA修复和重组过程上,如RuvA、RuvB、RuvC、RecG 和 LigA 等关键基因。进一步的结构分析发现,这些基因的关键部位(如DNA结合区和蛋白互动区)发生了特定变化。这样的分析流程简单直观,可以很容易拓展到寻找其它物种中对其特殊性状负责的相应基因上。
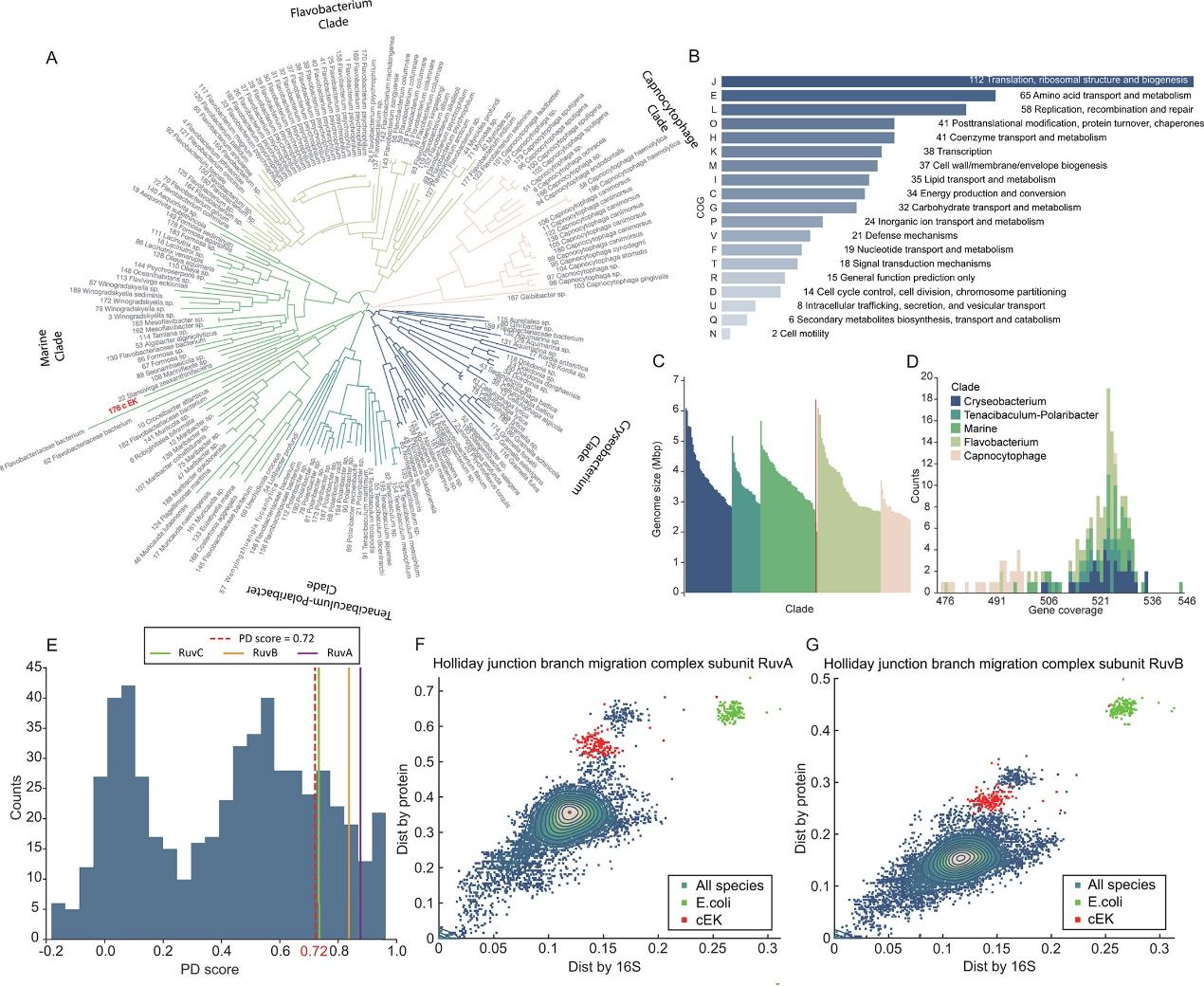
文中图2概述了基因组数据和PD分数计算, 包括197种物种的16S rRNA系统发育树(A)和RuvA(F)和RuvB(G)的蛋白距离 vs. 16S rRNA距离散点图,用于显示PD距离的直观理解。
实验验证:把“超级重组系统”移植进大肠杆菌
为了验证这些基因是否真能提升重组能力,研究团队将它们“搬家”到实验室常用菌株——E. coli(大肠杆菌)中。结果显示,这些海洋菌来源的基因让大肠杆菌的DNA重组效率显著提升,就像给它装上了“基因改造的涡轮引擎”。这不仅证明了 PD 方法的可靠性,也为未来提高基因编辑效率提供了可用工具。
意义:一把寻找“特长基因”的新钥匙
这项研究的意义在于提出了一种普适的“基因雷达”算法:它不依赖已知功能注释,也不限于孤儿基因,只要某个基因在进化图谱上“偏得出奇”,它就能被揪出来。
这让科学家有机会系统地探索更多有趣的性状,例如是什么基因让某些细菌能耐高温、抗辐射、或制造奇特化合物;以及如何借助这些“自然天赋”改进生物工程。
在未来,这一方法有望应用于发掘环境适应性基因、代谢创新基因,甚至高效DNA修复与重组模块等,为合成生物学、微生物进化学乃至生物技术产业提供新的“基因宝藏”。
该工作由国家重点研发计划(2024YFA0919500)、国家自然科学基金(T2321001、12090053 和 32088101)以及四川省科技计划项目(2025YFHZ0060)资助,并受到了生命联合中心的资金支持。
生命联合中心研究生周小琳、北大成都前沿交叉生物技术研究院李威杰和北大生科院本科生研究者谢思雅为该论文的共同第一作者。李志远和杨晓静为本文的共同通讯作者。
原文链接:https://journals.asm.org/doi/10.1128/mbio.03566-24
论文信息
Zhou X., Li W., Xie S., Xia K., Xiao M., Yang X., Li Z. A phylogeny-based method in detecting the species-specialized genes in microbes and its application to a marine symbiont bacterium. mBio (2025) DOI: 10.1128/mbio.03566-24