形态是细胞的基本几何与物理属性,能反映千变万化的生物学过程。例如,入侵性癌细胞的不规则形态反映其迁移能力,神经细胞的延展形态反映其连接能力。由于细胞密度、光毒性/光漂白性等限制,如何对完整的生命体系进行全面三维延时的细胞形态重构,是一个重要且具有挑战性的问题。此前,科学家曾在线虫(本研究团队于2020年的工作 [1])、海鞘 [2]、果蝇 [3]、斑马鱼 [4] 等物种进行部分胚胎发育过程的形态图谱构建,具备单细胞精度的全过程形态图谱仍未在任何物种里被构建 [5]。
近日,北京大学汤超实验室、香港浸会大学赵中应实验室、香港城市大学严洪实验室在Nature Communications在线发表文章Cell lineage-resolved embryonic morphological map reveals signaling associated with cell fate and size asymmetry [6][https://doi.org/10.1038/s41467-025-58878-0],系统构建了秀丽隐杆线虫(Caenorhabditis elegans,C. elegans)全胚胎发育过程的单细胞形态图谱(图1)。秀丽隐杆线虫拥有单细胞级别的发育精度,即每个细胞的分裂时间、分裂方向、运动路径、身份命运等信息在不同个体间高度准确,加上其丰富的遗传调控单元,秀丽隐杆线虫成为过去半个世纪内发育生物学界最为广泛使用的模式生物之一。1983年,Sulston J. E.(2002年诺贝尔生理医学奖)等人通过人工识别追踪,绘制出线虫胚胎发育的单细胞时空谱系 [7];2006年,Waterston R. H.等人开发了持续标记、追踪全胚胎细胞核的计算框架 [8]。然而,细胞核只是细胞体内一个点,无法准确提供复杂且重要的三维形态信息;而细胞膜在空间中呈区域连续分布,比细胞核更难识别。本研究重点扩展发育图谱的细胞形态维度(图1)。
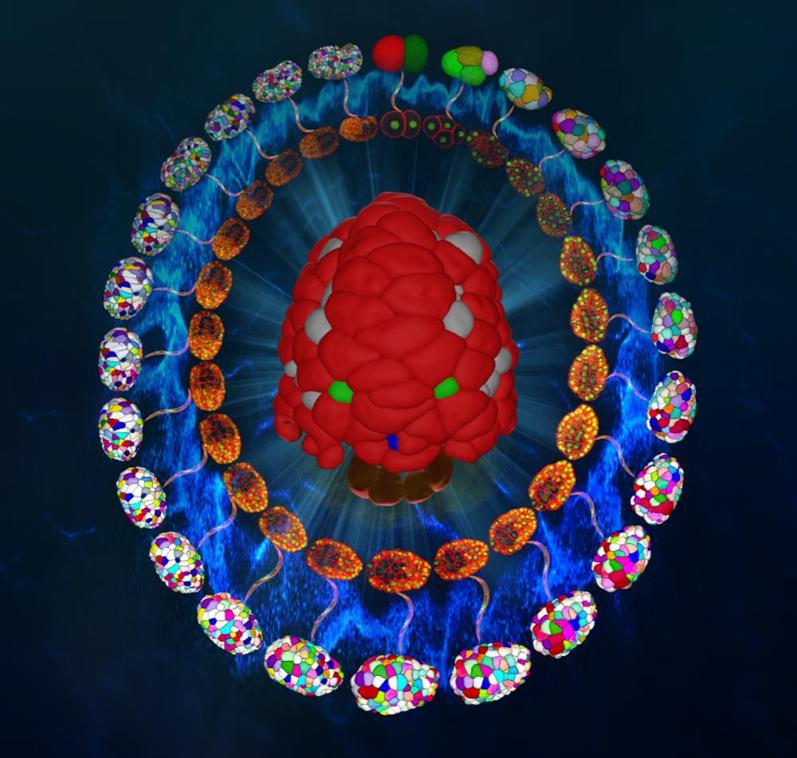
图1. 秀丽隐杆线虫从受精到伸长期的荧光拍摄与形态重构。
1. 全胚胎发育过程形态图谱的定量构建
研究团队首先对线虫胚胎以小于1.5分钟的时间间隔进行三维拍摄,拍摄时间不少于6小时,其中细胞核被绿色荧光标记(用于细胞身份追踪),细胞膜被红色荧光标记(用于细胞形态重构)。最终研究团队获得一共8个野生型胚胎、共约40万个独立的三维细胞区域,覆盖从受精后第一次分裂完成到胚胎伸长期,记录超过95%的胚胎细胞。伴随的定量信息包括细胞身份、谱系、命运、形状、体积、表面积、接触关系与接触面积。该延时三维数据能够被用来定性和定量刻画不同谱系、组织器官的形态动力学(图2、图3)。
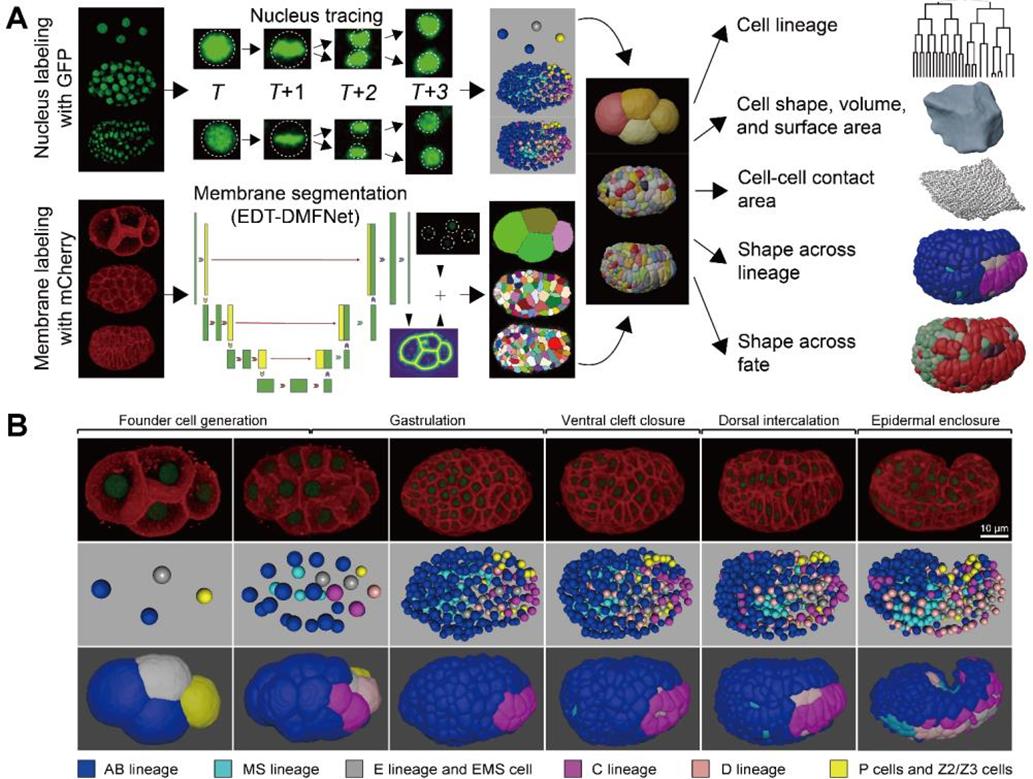
图2. 线虫不同谱系的单细胞精度形态重构与数据定量流程。
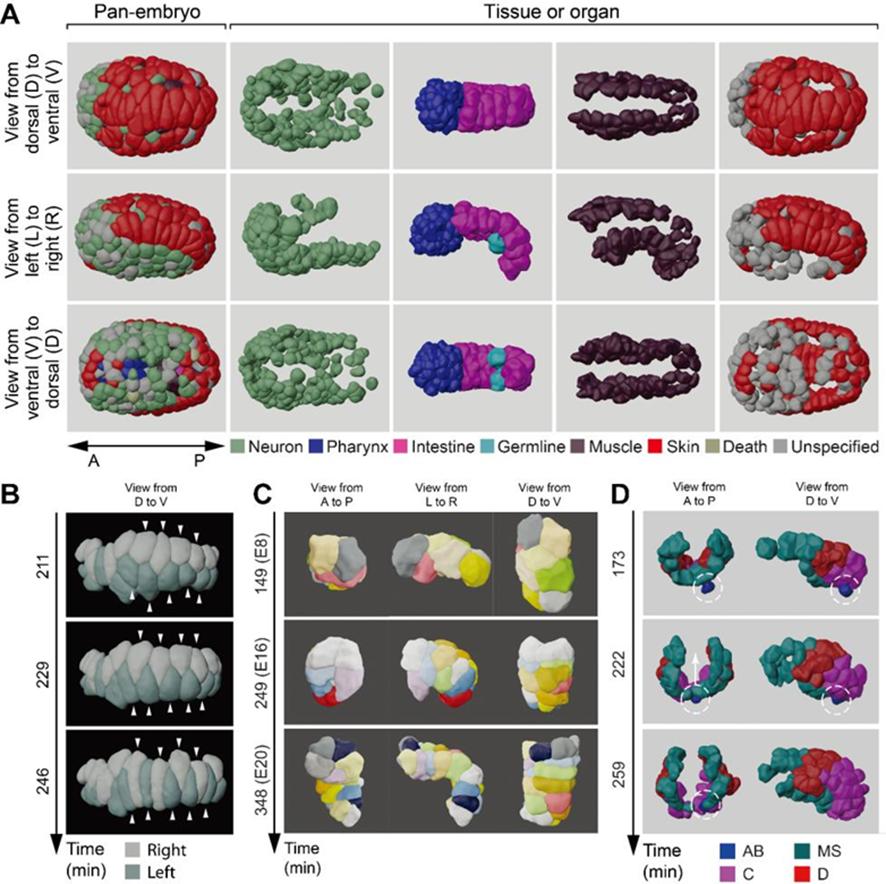
图3. 线虫不同组织器官(神经、咽喉、肠道、性腺、肌肉、皮肤等)的单细胞精度形态重构。
2. 细胞不对称分裂的信号调控机制
细胞形态常常以多种功能特征参与到发育过程,比如以Notch为代表的邻居细胞信号转导整合细胞接触关系和接触面积,诱导后续咽喉、头部、肾脏的分化 [9]。因此,研究团队进一步从细胞形态数据提取体积、表面积、接触关系与接触面积,继续考察Notch信号如何控制细胞分化。研究团队参考前人报道的Notch配对细胞,发现接收信号(或更强信号)的细胞在分裂过程中会产生更大的头部子代细胞。这种体轴不对称性在6组配对细胞里毫无例外,并通过信号阻断实验得以独立验证(图4)。
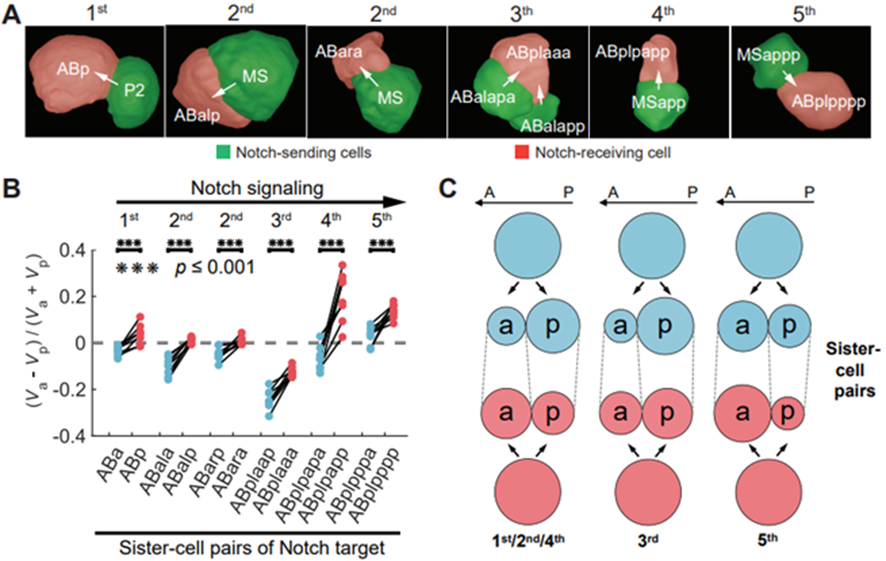
图4. Notch信号转导使细胞分裂获得更强的头尾不对称性。
研究团队继续好奇该体积不对称性是否可能具备生理学功能。此前线虫第4次Notch信号已经被发现对肾脏细胞(单细胞器官)在分子层面上起诱导分化作用,该单细胞在成体内具备显著的大体积并贯穿个体 [9]。研究团队发现除过去报道的信号接收细胞以外,其后续两轮分裂依然受信号影响,从而通过多轮不对称分裂使最终的肾脏细胞成为AB谱系(首个体细胞谱系)内最大的细胞(图5)。
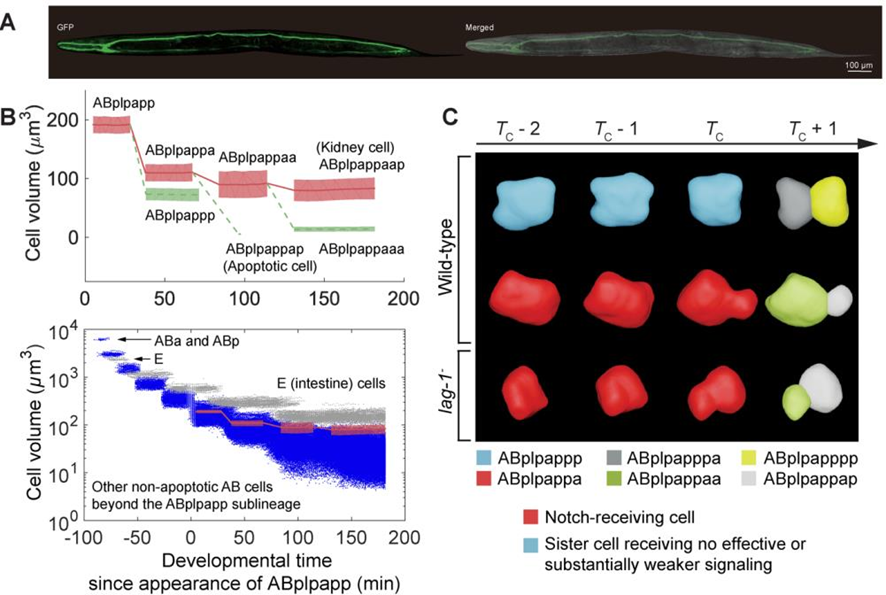
图5. Notch信号转导使肾脏细胞谱系获得多轮不对称分裂与最终显著的大体积。
3. 基因表达与细胞形态数据的整合分析
研究团队进一步从以往文献和新培养的线虫胚胎中收集超过400个基因的单细胞表达图谱。本文章数据可以通过ITK-SNAP-CVE(C. elegans virtual embryogenesis;https://doi.org/10.6084/m9.figshare.24768921)软件和CMOS(cellular morphology of C. elegans embryo; https://bcc.ee.cityu.edu.hk/cmos)网站访问(图6、图7)。研究团队成功结合Notch信号配体/受体表达谱、细胞体积、细胞接触识别出5组新的信号配对,并通过信号阻断和激光销蚀实验独立验证。
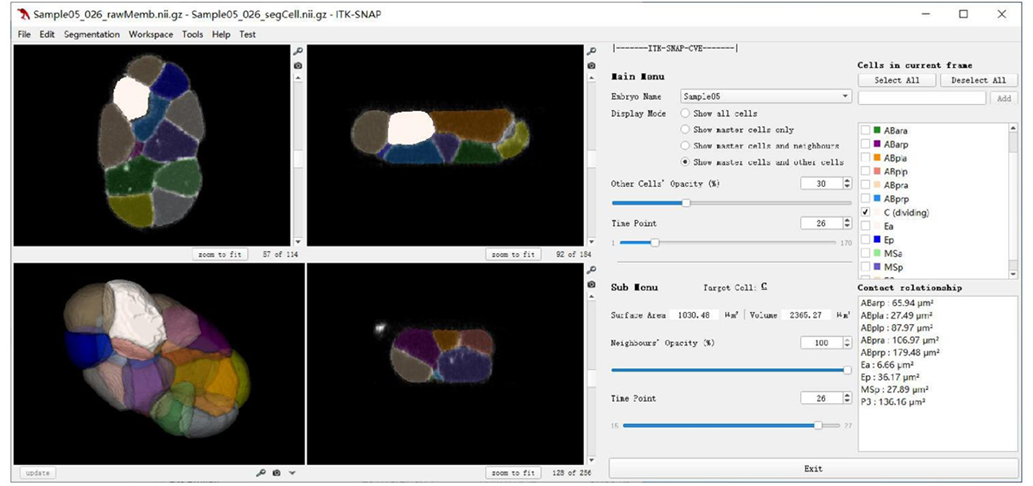
图6. ITK-SNAP-CVE软件平台。
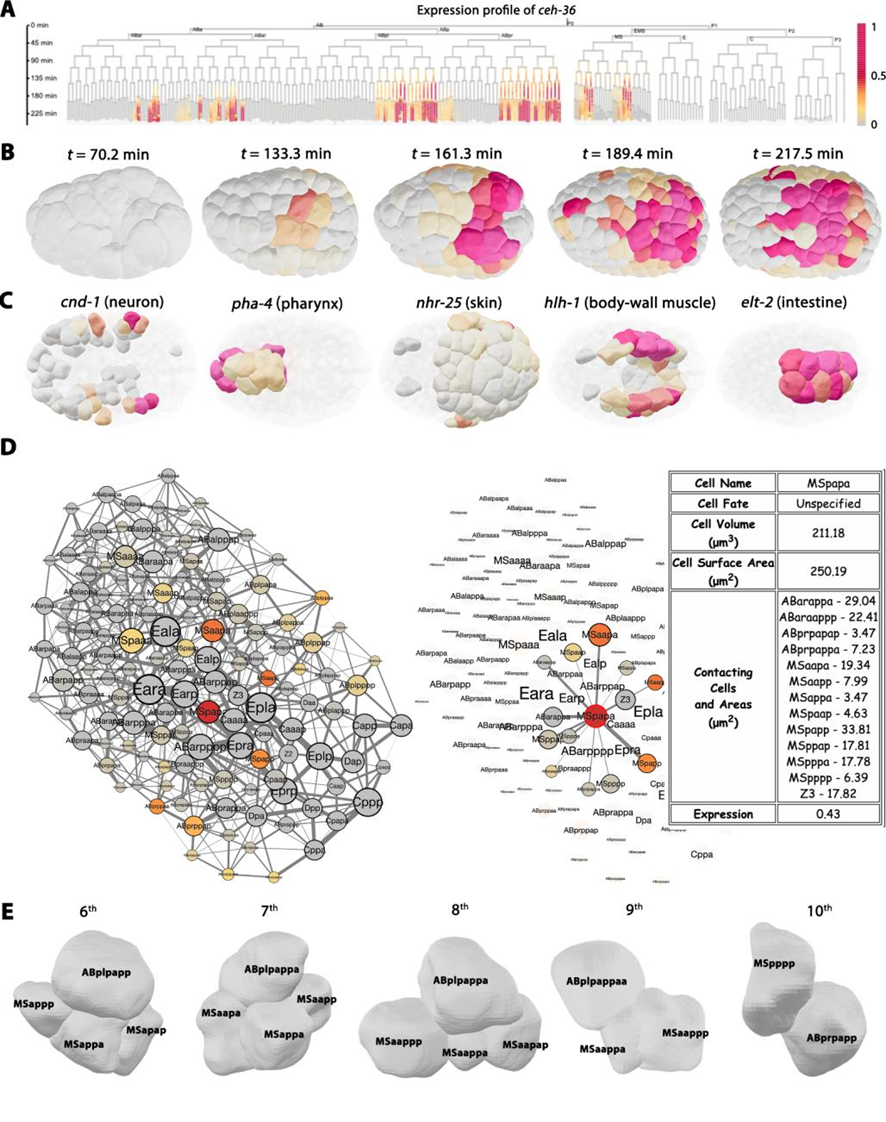
图7. CMOS网页平台。
4. 总结与展望
单细胞精度的时空、多维数据获取存在技术难度,特别是对于高分辨率、长拍摄时长等要求下的生命过程。本研究团队在多学科交叉融合的背景下,长期在实验显示手段 [10-13]、计算机视觉 [14-17]、定量发育数据分析 [18-21] 等方面推进多年,于2020年完成线虫胚胎形态图谱前半部分 [1],而在本工作完成后半部分,并兼容力学挤压、突变体、RNA干扰体、激光销蚀等实验条件。本工作提供的高质量生物数据可以被用于物理模型发展和生物原理挖掘 [21-24]、细胞形态与基因/命运/分化的关联识别 [26-27]、以及为机器学习/人工智能(AI for Science)提供充足的数据基础。
北京大学定量生物学中心/生命科学联合中心/物理学院(现西湖大学交叉科学中心)汤超教授、香港浸会大学生物系赵中应教授、香港城市大学电子工程系/智能多维数据分析研究中心有限公司严洪教授为文章的共同通讯作者;北京大学博士生关国业(现哈佛医学院/丹娜法伯癌症研究院博士后)、香港城市大学博士生李泽霖、香港浸会大学博士生马一鸣、香港浸会大学博士生叶珀豪为文章的共同第一作者;原香港城市大学曹剑锋博士提供了计算支持;原香港浸会大学Vincy Wing Sze Ho博士、Ming-Kin Wong博士、Lu-Yan Chan提供了实验支持。

部分团队成员在香港共进早餐,拍摄于2023年夏天
5. 文献
1) Cao†, Guan†, Ho†, et al. Establishment of a morphological atlas of the Caenorhabditis elegans embryo using deep-learning-based 4D segmentation. Nat. Commun. (2020)
2) Guignard†, Fiúza†, et al. Contact area-dependent cell communication and the morphological invariance of ascidian embryogenesis. Science (2020)
3) Khan†, et al. Quantitative 4D analyses of epithelial folding during Drosophila gastrulation. Development (2014)
4) Stegmaier†, et al. Real-time three-dimensional cell segmentation in large-scale microscopy data of developing embryos. Dev. Cell (2016)
5) Guan†, et al. Delineating the mechanisms and design principles of Caenorhabditis elegans embryogenesis using in toto high-resolution imaging data and computational modeling. Comput. Struct. Biotechnol. J. (2022)
6) Guan†, Li†, Ma†, Ye†, et al. Cell lineage-resolved embryonic morphological map reveals signaling associated with cell fate and size asymmetry. Nat. Commun. (2025)
7) Sulston†, et al. The embryonic cell lineage of the nematode Caenorhabditis elegans. Dev. Biol. (1983)
8) Bao†, et al. Automated cell lineage tracing in Caenorhabditis elegans. Proc. Natl. Acad. Sci. U. S. A. (2006)
9) Priess†, J. R. Notch signaling in the C. elegans embryo. WormBook (2005)
10) Chen†, Ho†, Wong†, Huang†, et al. Establishment of signaling interactions with cellular resolution for every cell cycle of embryogenesis. Genetics (2025)
11) Cao†, et al. 3DMMS: Robust 3D membrane morphological segmentation of C. elegans embryo. BMC Bioinform. (2019)
12) Wong†, Ho†, et al. Initial characterization of gap phase introduction in every cell cycle of C. elegans embryogenesis. Front. Cell Dev. Biol. (2022)
13) Li†, Xie†, Ma†, et al. Deep learning-based enhancement of fluorescence labeling for accurate cell lineage tracing during embryogenesis. Bioinformatics (2024)
14) Li†, et al. 3D cellular segmentation of live embryos via topologically and biologically boundary-aware semi-supervised learning. 2024 IEEE International Conference on Systems, Man, and Cybernetics (2024)
15) Li†, et al. Directional and topological transformer with topology priors for 4D cellular image segmentation. 2024 IEEE International Conference on Image Processing (2024)
16) Huang†, et al. A fusion model with effective multi-scale parallel transformer for cellular segmentation. 2025 IEEE Transactions on Computational Biology and Bioinformatics (2025)
17) Li†, et al. Volume tells: Dual cycle-consistent diffusion for 3D fluorescence microscopy de-noising and super-resolution. 2025 Conference on Computer Vision and Pattern Recognition (2025)
18) Huang†, Zhu†, et al. An integrative C. elegans protein-protein interaction network with reliability assessment based on a probabilistic graphical model. Mol. BioSyst. (2016)
19) Huang†, Zhu†, et al. Inference of cellular level signaling networks using single-cell gene expression data in Caenorhabditis elegans reveals mechanisms of cell fate specification. Bioinformatics (2016)
20) Guan†, Fang†, et al. Multilevel regulation of muscle-specific transcription factor hlh-1 during Caenorhabditis elegans embryogenesis. Dev. Genes Evol. (2020)
21) Guan†, et al. Investigating spatio-temporal cellular interactions in embryonic morphogenesis by 4D nucleus tracking and systematic comparative analysis. 2021 IEEE 9th International Conference on Bioinformatics and Computational Biology (2021)
22) Guan†, et al. Volume segregation programming in a nematode's early embryogenesis. Phys. Rev. E (2021)
23) Kuang†, Guan†, et al. Computable early Caenorhabditis elegans embryo with a phase field model. PLoS Comput. Biol. (2022)
24) Guan†, Kuang†, et al. Comparison between phase-field model and coarse-grained model for characterizing cell-resolved morphological and mechanical properties in a multicellular system. Commun. Nonlinear Sci. Numer. Simul. (2023)
25) Kuang†, Guan†, et al. MorphoSim: An efficient and scalable phase-field framework for accurately simulating multicellular morphologies. npj Syst. Biol. Appl. (2023)
26) Li†, et al. 3DCSQ: An effective method for quantification, visualization and analysis of 3D cell shape during early embryogenesis. Quant. Biol. (2024)
27) Guan†, Chen†, et al. Characterizing cellular physiological states with 3D shape descriptors for cell membranes. Membranes (2024)